Observer en détail le cerveau humain intact, quelle que soit sa taille, a longtemps été un rêve inatteignable pour les neurosciences. Cependant, une nouvelle étude publiée dans Science par une équipe du MIT présente une avancée significative. Ils ont développé une technologie permettant de traiter, d’étiqueter et d’imager avec précision des hémisphères cérébraux entiers de deux donneurs – l’un atteint de la maladie d’Alzheimer et l’autre non – avec une haute résolution et rapidité.
« Nous avons réalisé une imagerie holistique des tissus cérébraux humains à plusieurs résolutions, depuis des synapses uniques jusqu’à des hémisphères cérébraux entiers, et nous avons rendu ces données disponibles », déclare Kwanghun Chung, professeur agrégé au MIT. « Ce pipeline technologique nous permet d’analyser le cerveau humain à plusieurs échelles et pourrait potentiellement être utilisé pour cartographier entièrement le cerveau humain. »
Bien que l’étude ne présente pas une carte complète du cerveau, elle démontre une suite intégrée de trois technologies permettant une imagerie hémisphérique complète. Cette recherche fournit une « preuve de concept » en montrant ce que le pipeline rend possible, notamment des vues de milliers de neurones, des forêts de cellules avec des détails individuels, et des structures subcellulaires nichées parmi les molécules extracellulaires. Les chercheurs ont également effectué des comparaisons analytiques quantitatives entre les hémisphères atteints et non atteints d’Alzheimer.
L’importance de pouvoir imager des hémisphères entiers du cerveau humain intact jusqu’à la résolution des synapses individuelles est cruciale pour comprendre le cerveau en bonne santé et en maladie, explique Chung.
Un cerveau vaut mieux que deux
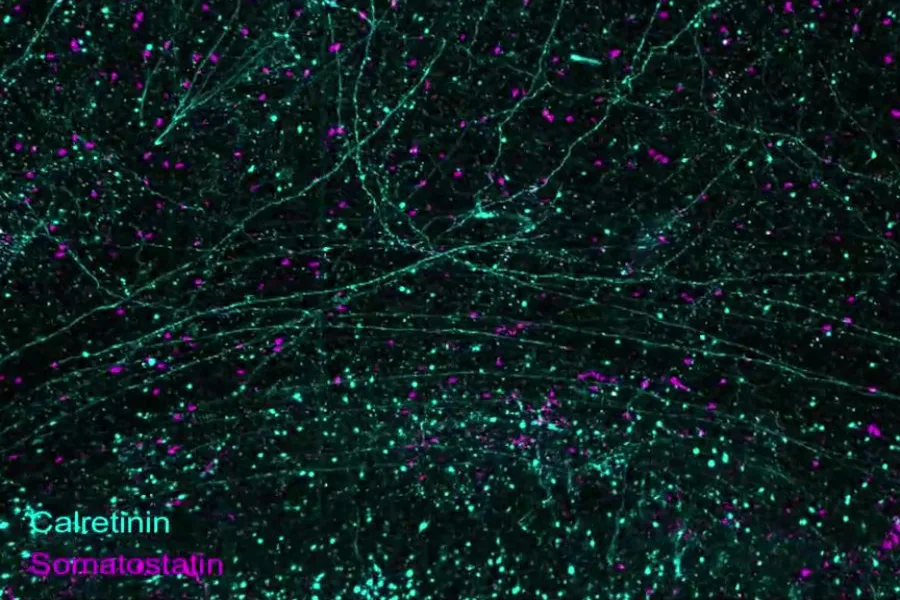
Cette technologie permet aux scientifiques d’explorer diverses questions en utilisant le même cerveau, évitant ainsi les variations entre différents cerveaux. Une caractéristique clé est que l’analyse ne dégrade pas les tissus, les rendant extrêmement durables et ré-étiquetables pour de nouvelles études pendant des années. L’équipe a démontré l’utilisation de 20 marqueurs d’anticorps différents et prévoit d’étendre ce chiffre à une centaine ou plus.
« Nous devons être capables de voir tous ces différents composants fonctionnels – les cellules, leur morphologie et leur connectivité, leurs architectures subcellulaires et leurs connexions synaptiques individuelles – idéalement au sein du même cerveau », explique Chung. « Ce pipeline technologique nous permet d’extraire toutes ces fonctionnalités importantes de manière totalement intégrée. »
Le pipeline est également évolutif et rapide (l’imagerie d’un hémisphère cérébral entier prend 100 heures). Chung envisage de créer une banque de cerveaux entièrement imagés pour permettre des comparaisons robustes avec une puissance statistique accrue.
Trois innovations clés
Chung a constitué une équipe au MIT comprenant trois jeunes scientifiques talentueux, chacun co-auteur principal de l’article. Ji Wang a développé le « Mégatome », un appareil permettant de trancher finement les hémisphères du cerveau humain sans les endommager. Juhyuk Park a développé la chimie rendant chaque tranche de cerveau claire, flexible, durable et étiquetable rapidement – une technologie appelée « mELAST ». Webster Guan a créé un système informatique appelé « UNSLICE » pour reconstruire chaque hémisphère en 3D.
Aucune technologie ne permet d’imager l’anatomie entière du cerveau humain à une résolution subcellulaire sans le découper au préalable. Mais le Megatome de Wang permet de trancher rapidement et finement les hémisphères sans perdre d’information anatomique. Park a conçu un hydrogel qui rend les échantillons de cerveau optiquement clairs et pratiquement indestructibles. Guan a développé UNSLICE pour restaurer une image intacte de l’hémisphère par calcul.
Dans l’étude, les chercheurs présentent divers exemples de ce que le pipeline peut faire, notamment l’étiquetage riche d’un hémisphère entier et le zoom sur des structures cérébrales, des circuits, des cellules individuelles et des composants subcellulaires.
Explorer la maladie d’Alzheimer
Chung a collaboré avec Matthew Frosch pour imager et comprendre les cerveaux atteints de la maladie d’Alzheimer. Ils ont exploré les régions du cerveau présentant une perte neuronale importante et ont utilisé de nombreux marqueurs pour observer les relations entre les facteurs pathogènes et les différents types de cellules.
« Ce pipeline nous permet d’avoir un accès presque illimité aux tissus », explique Chung. « Nous pouvons toujours revenir en arrière et envisager quelque chose de nouveau. »
Ils ont concentré leur analyse sur le cortex orbitofrontal de chaque hémisphère, observant que la perte de synapses était concentrée dans les zones de chevauchement avec les plaques amyloïdes.
Avec seulement deux échantillons, l’équipe ne propose aucune conclusion sur la nature de la maladie d’Alzheimer, mais l’étude montre qu’il est désormais possible d’imager et d’analyser en profondeur des hémisphères entiers du cerveau humain pour permettre ce genre de recherche.
Notamment, la technologie s’applique également à de nombreux autres tissus du corps, pas seulement au cerveau.
« Nous envisageons que cette plate-forme technologique évolutive fera progresser notre compréhension des fonctions des organes humains et des mécanismes des maladies afin de stimuler le développement de nouvelles thérapies », concluent les auteurs.
Outre Park, Wang, Guan, Chung et Frosch, les autres auteurs de l’article sont Lars A. Gjesteby, Dylan Pollack, Lee Kamentsky, Nicholas B. Evans, Jeff Stirman, Xinyi Gu, Chuanxi Zhao, Slayton Marx, Minyoung E. Kim, Seo Woo Choi, Michael Snyder, David Chavez, Clover Su-Arcaro, Yuxuan Tian, Chang Sin Park, Qiangge Zhang, Dae Hee Yun, Mira Moukheiber, Guoping Feng, X. William Yang, C. Dirk Keene, Patrick R. Hof, Satrajit S. Ghosh et Laura J. Brattain.
Le financement principal du travail provenait des National Institutes of Health, du Picower Institute for Learning and Memory, de la JPB Foundation et de la NCSOFT Cultural Foundation.